دنباله موتیف
در ژنتیک یک دنباله موتیف الگویی از توالی نوکلئوتید یا آمینو اسید است. برای پروتئینها توالی موتیف از طریق ساختار سهبعدی آمینو اسیدهای تشک.
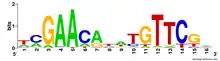
مثال زیر یک موتیف N-گلیکوزیلاسیون است:
- یک Asn به دنبال آن هر چیزی بجز Pro میتواند بیاید و پس از آن یک Ser یا Thr خواهد آمد. در ادامه در چیزی بجز Pro میتواند باشد.
عبارتهای سهحرفی آمده در بالا نامهای اختصاری آمینواسیدها هستند.
بررسی اجمالی
زمانی که یک دنباله موتیف در اگزون یک ژن ظاهر میشود، ممکن است بهصورت رمز "ساختاری موتیف" از یک پروتئین تفسیر گردد; که یک کلیشه عنصر از ساختار کلی از پروتئین است. با این وجود الزاماً موتیف به یک ثانویه ساختار خاص وابسته نیست. توالیهای "Noncoding" به پروتئین ترجمه نمیشوند.
خارج از بخش اگزون ژنها، دنباله موتیفهای نظارتی وجود دارند. برخی از اینها بر شکل اسیدهای نوکلئیک (نگاه کنید به عنوان مثال RNA خود پیرایش) تاثیر میگدارند اما این تنها در مورد برخی از موارد صدق میکند. برای مثال بسیاری از پروتئینهای متصل شونده به DNA دارای شکل خاصی هستند که باعث میشود بتوانند موتیفها را در DNA تشخیص دهند و در آن نواحی به DNAمتصل شوند.
پژوهشگران برای یافتن موتیفها از اطلاعات از ابزارهای موجود در بیوانفورماتیک که برای تحلیل اطلاعات کاربرد دارند (مانند BLAST) استفاده میکنند.
نمایش موتیف
مثال زیر را که یک موتیف N-گلیکوزیلاسیون است، در نظر بگیرید:
- یک Asn به دنبال آن هر چیزی بجز Pro میتواند بیاید و پس از آن یک Ser یا Thr خواهد آمد. در ادامه در چیزی بجز Pro میتواند باشد.
این الگو را میتوان به صورت
N{P}[ST]{P}
نمایش میدهند که در آن
N = Asn, P = Pro, S = Ser, T = Thr;
است.
عبارت {X} به این معنی است که هر آمینواسیدی بجز X میتواند در این مکان قرار بگیرد. همچنین عبارت [XY] یعنی در این مکان یکی از آمینواسیدهای X یا Y میتواند قرار بگیرد. مشکل نشانگذاری [XY] این است که در آن نمیتوان احتمال حضور هر کدام از آمینواسیدها را در این مکان نشان داد. بدین منظور میتوان از نمایش sequence logo استفاده کرد.
الگوی نمایش دنباله موتیف
روشهای مختلفی برای نمایش دنباله موتیف وجود دارد، اما اکثر این روشها از روش استاندارد عبارت باقاعده با قراردادهای زیر استفاده میکنند:
- نمادگذاری از حروف برای نمایش آمینواسیدها وجود دارد که در آن هر بک حرف نماد یک آمینواسید است.
- دنبالهای از حروف نشان دهندهی دنبالهای از آمینواسیدهای مربوط به هر حرف است.
- به ازای دنبالهای از حروف که داخل براکت قرار گرفتهاند، تنها یک آمینواسید که حرف آن در رشتهی داخل براکت ذکر شده، میتواند بیان شود. برای مثال : بهجای
[abc]تنها آمینواسید متناظر باaیاbیاcمیتواند بیان شود.
ماتریس
یک ماتریس عددی حاوی امتیازهای هر نوکلئوتید در هر موقعیت از یک موتیف با طول ثابت است. دو نوع ماتریس وزندهی وجود دارد.
- یک ماتریس فرکانس موقعیت (PFM)، ثبت فرکانس وابسته به موقعیت هر یک از نوکلئوتید است. PFMs میتواند به صورت تجربی از آزمایش SELEX تعیین گردد یا با محاسباتی توسط ابزارهایی مانند الگوهای رفتاری با استفاده از مدل مخفی مارکوف تعیین گردد.
- یک ماتریس وزن موقعیت (PWM) شامل لگاریتم شانس وزندار انطباق است.یک حد آستانه نیز مورد نیاز است تا مشخص کند که آیا یک دنباله ورودی موتیف با الگوی ما منطبق است یا نه. PWM از PFM محاسبه می شود.
نمونه ای از یک PFM از پایگاه داده TRANSFAC برای فاکتور رونویسی AP-1:
| Pos | A | C | G | T | IUPAC |
|---|---|---|---|---|---|
| 01 | 6 | 2 | 8 | 1 | R |
| 02 | 3 | 5 | 9 | 0 | S |
| 03 | 0 | 0 | 0 | 17 | T |
| 04 | 0 | 0 | 17 | 0 | G |
| 05 | 17 | 0 | 0 | 0 | A |
| 06 | 0 | 16 | 0 | 1 | C |
| 07 | 3 | 2 | 3 | 9 | T |
| 08 | 4 | 7 | 2 | 4 | N |
| 09 | 9 | 6 | 1 | 1 | M |
| 10 | 4 | 3 | 7 | 3 | N |
| 11 | 6 | 3 | 1 | 7 | W |
ستون اول موقعیت را مشخص میکند، ستون دوم شامل تعداد وقوع A در آن موقعیت است ، ستون سوم شامل تعداد تکرار C در آن موقعیت است، ستون چهارم شامل تعداد تکرار G در آن موقعیت است، ستون پنجم شامل تعداد تکرار T در آن موقعیت میباشد، و آخرین ستون شامل نماد IUPAC برای آن موقعیت. توجه داشته باشید که جمع تعداد وقوع A، C، G، T برای هر ردیف باید با ردیفهای دیگر یکسان باشد.
مدل کدگذاری
مثال زیر از مقالهای از ماتسودا و همکاران که در سال 1997 منتشر شده است [1]، گرفته شده است:
ماتسودا، و همکاران. یک کدگذاری به نام "کد های زنجیره ای سه بعدی" برای ارائه یک ساختار پروتئین به عنوان یک رشته از حروف نام پیشنهاد دادند. این طرح شباهت بین پروتئینها را بسیار واضحتر نسبت به توالی اسید آمینهها نشان می دهد:
| 3D زنجیرهی کد | توالی اسید آمینه | |
|---|---|---|
| 1lccA | TWWWWWWWKCLKWWWWWWG | LYDVAEYAGVSYQTVSRVV |
| 3gapA | KWWWWWWGKCFKWWWWWWW | RQEIGQIVGCSRETVGRIL |
که در آن "W" مربوط به یک α-مارپیچ و "E" و "د" مربوط به یک β-رشته است.
منابع
- Matsuda H; Taniguchi F; Hashimoto A (1997). "An approach to detection of protein structural motifs using an encoding scheme of backbone conformations" (PDF). Proc. of 2nd Pacific Symposium on Biocomputing: 280–291. Archived from the original (PDF) on 5 March 2012. Retrieved 30 December 2016.
مطالعه بیشتر
- Stormo GD (2000). "DNA binding sites: representation and discovery". Bioinformatics. 16 (1): 16–23. doi:10.1093/bioinformatics/16.1.16. PMID 10812473.
- Balla S; Thapar V; Verma S; Luong T; Faghri T; Huang CH; Rajasekaran S; del Campo JJ; Shinn JH; Mohler WA; Maciejewski MW; Gryk MR; Piccirillo B; Schiller SR; Schiller MR (2006). "Minimotif Miner: a tool for investigating protein function". Nature Methods. 3 (3): 175–177. doi:10.1038/nmeth856. PMID 16489333.
- Schiller MR (2007). "Minimotif miner: a computational tool to investigate protein function, disease, and genetic diversity". Curr Protoc Protein Sci. chapter 2 (unit 2.12): Unit 2.12. doi:10.1002/0471140864.ps0212s48. PMID 18429315.
- Kadaveru K; Vyas J; Schiller MR (2008). "Viral infection and human disease--insights from minimotifs". Front Biosci. 13 (13): 6455–6471. doi:10.2741/3166. PMC 2628544. PMID 18508672.
- Doaa Altarawy; M. A. Ismail & Sahar Ghanem (2009). "MProfiler: A Profile-Based Method for DNA Motif Discovery". Pattern Recognition in Bioinformatics. 5780: 13–23. doi:10.1007/978-3-642-04031-3_2.