یادگیری ماشین در بیوانفورماتیک
با پیشرفت تکنولوژی و افزایش چشمگیر دادههای زیستی، علاوه بر ذخیرهسازی و نگهداری، استخراج اطلاعات سودمند از این حجم از داده نیز چالش بزرگی را برای پژوهشگران به وجود آوردهاست. به این منظور، برای به دست آوردن دانش از دادههای زیستی از ابزارها و روشهای یادگیری ماشین استفاده میشود.[1] یادگیری ماشین که زیرشاخهای از علوم رایانه است، دارای کاربردهای بسیاری در بیوانفورماتیک است. بیوانفورماتیک دانشی است که به جنبههای ریاضی و محاسباتی زیستشناسی برای فهم و پردازش دادههای زیستی میپردازد.[2]
پیش از ظهور روشهای یادگیری ماشین در بیوانفورماتیک، الگوریتمهای بیوانفورماتیک به صورت دستنویس و غیرخودکار برنامهنویسی میشدند، که برای مسائلی مانند پیشبینی ساختار پروتئین بسیار دشوار بودهاست. روشهایی در یادگیری ماشین مانند یادگیری عمیق به الگوریتم این اجازه را میدهد که از روی ویژگیهای اولیهٔ دادهٔ ورودی ویژگیهایی پیچیدهتر را برای بهکارگیری در الگوریتم یادگیری بسازد. این نوع سیستمها با داشتن حجم بزرگی از داده برای یادگیری میتوانند پیشبینیهای کاملاً پیچیدهای را انجام دهند. در سالهای اخیر حجم دادههای زیستی به شدت افزایش یافتهاست، که این موضوع استفاده از سیستمهای گفته شده را برای پژوهشگران بیوانفورماتیک میسر میکند.[2]
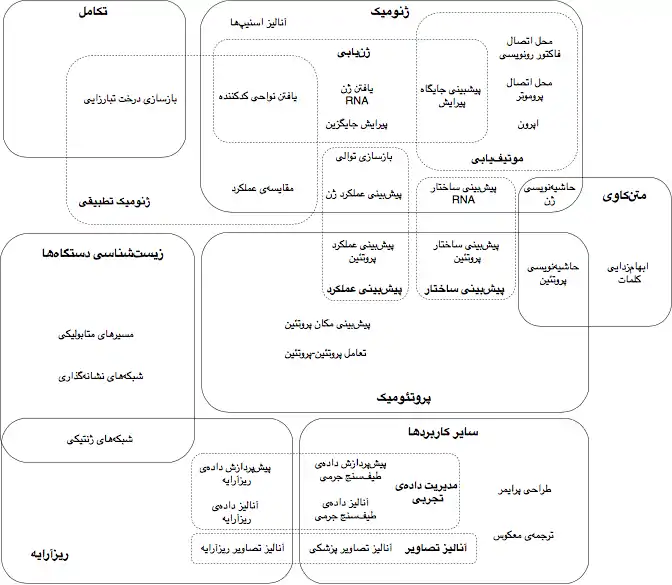
یادگیری ماشین در شش شاخه از زیستشناسی مورد استفاده قرار میگیرد. این شاخهها عبارتند از: ژنومیک، پروتئومیک، ریزآرایه، زیستشناسی دستگاهها، تکامل و متنکاوی.
کاربردها
ژنومیک

ژنومیک شامل مطالعهٔ ژنوم موجودات زنده، که رشتهٔ دیانای کامل آنها است، میشود. با وجود اینکه دادههای ژنومیک به دلیل مشکلات فنی در توالییابی یک قطعه از دیانای در طول زمان ناقص بودهاست، تعداد رشتههای موجود به صورت نمایی در حال افزایش است. برخلاف افزایش نمایی این نوع از دادهٔ خام، تفسیر زیستی آن با سرعت بسیار کمتری انجام میشود. به این دلیل به روشهای یادگیری ماشین برای تشخیص محل ژنهایی که به پروتئین ترجمه میشوند، روی آورده میشود. این مسئله به مسألهٔ ژنیابی معروف است.
علاوه بر مسألهٔ ژنیابی، روشهای یادگیری ماشین در مسألهٔ همترازسازی چند توالی نیز استفاده میشود. در این مسئله، تعداد زیادی رشتهٔ دیانای یا اسید آمینه به منظور یافتن نواحی مشابه همترازسازی میشوند. این نواحی مشابه میتوانند نشاندهندهٔ اطلاعاتی در مورد پیشزمینهٔ تکاملی این رشتهها باشند.[2]
پروتئومیک
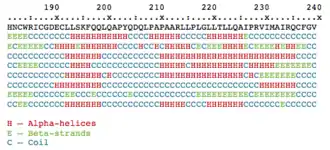
پروتئینها که رشتههایی متشکل از اسید آمینهها هستند، بخش بزرگی از کارایی و عملکرد خود را از تاشدگی میگیرند که به آنها ساختاری سهبعدی میدهد. این ساختار شامل ۴ لایه که به آنها ساختار اول تا چهارم گفته میشود، میشود. ساختار اولیهٔ پروتئین نشاندهندهٔ توالی اسید آمینهها است و ساختار دوم آن شامل مارپیچهای آلفا و صفحات بتا میشود.
از آنجایی که ساختارهای سوم و چهارم وابستگی زیادی به ساختار دوم دارند، در زیرشاخهٔ پروتئومیک توجه زیادی به ساختار دوم میشود. به دست آوردن ساختار کامل و دقیق پروتئین فرایندی بسیار پیچیده و زمانگیر است. پیش از استفاده از روشهای یادگیری ماشین، پژوهشگران سیستمهای پیشبینی ساختار پروتئین را به صورت دستی پیادهسازی میکردند. امروزه روشهای یادگیری ماشین با به دست آوردن خودکار ویژگیهای داده به دقت ۸۴٪-۸۲ رسیدهاند. در حال حاضر الگوریتم سرآمد در حوضهٔ پیشبینی ساختار دوم از سیستمی به نام DeepCNF استفاده میکند که بر اساس مدل شبکهٔ عصبی مصنوعی، به دقت ۸۴٪ در دستهبندی اسید آمینههای یک رشتهٔ پروتئین به دستههای مارپیچ، صفحه و سیمپیچ رسیدهاست. از لحاظ تئوری، حداکثر دقت قابل دستیابی در این مسئله برابر ۹۰٪-۸۸ است.
روشهای یادگیری ماشین در مسئلههای دیگری مانند پیشبینی زنجیر جانبی و مدل کردن خمیدگیهای پروتئین نیز استفاده میشوند.[2]
ریزآزایه
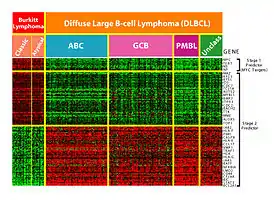
ریزآرایه یکی از انواع آزمایشگاه روی تراشه است که برای جمعآوری داده از مواد زیستی با مقدار بالا استفاده میشود. یادگیری ماشین میتواند در آنالیز این نوع داده کمک کند و در مواردی مانند مشخص کردن الگوهای بیان ژنها، دستهبندی و استنتاج از شبکههای ژنتیکی مورد استفاده قرار گیرد.
این تکنولوژی بهطور ویژه برای نظارت بر بیان ژنهای یک ژنوم به منظور تشخیص انواع مختلف سرطان استفاده میشود. یکی از مهمترین مسئلهها در این عرصه تشخیص ژنهایی است که بیان شدهاند. حجم بالای داده و وجود دادههای بیربط، این مسئله را سختتر میکند. روشهای دستهبندی در یادگیری ماشین مانند شبکهٔ تابع پایه شعاعی، یادگیری عمیق، دستهبندیکنندهٔ بیزی، درخت تصمیم و جنگل تصادفی در این مسئله استفاده میشوند.[2]
زیستشناسی دستگاهها
زیستشناسی دستگاهها به بررسی رفتارهای شدید فعل و انفعالات پیچیده در بین اجزاء سادهٔ زیستی میپردازد. چنین اجزائی میتوانند شامل مولکولهایی مانند دیانای، آرانای، پروتئینها و متابولیتها شوند.
یادگیری ماشین در مدل کردن فعل و انفعالات پیچیده در سیستمهایی مانند شبکههای ژنتیکی، شبکههای انتقال سیگنال و مسیرهای متابولیکی به کار میآید. مدلهای گرافیکی احتمالاتی یکی از پرکاربردترین روشها در مدل کردن شبکههای ژنتیکی هستند. علاوه بر این از روش بهینهسازی زنجیره مارکوف نیز در مسئلههایی مانند تشخیص نواحی اتصال فاکتور رونویسی استفاده میشود. الگوریتمهای ژنتیکی که روشهایی بر اساس روند طبیعی تکامل هستند در مدل کردن شبکههای ژنتیکی و ساختارهای تنظیمکننده مورد استفاده قرار میگیرند.
کاربردهای دیگر یادگیری ماشین در زیستشناسی دستگاهها عبارتند از: پیشبینی عملکرد آنزیمها، آنالیز دادهٔ ریزآرایههای توان بالا و پیشبینی عملکرد پروتئین.[2]
تکامل
در علم تکامل، به خصوص در بازسازی درخت تبارزایی نیز از روشهای یادگیری ماشین استفاده میشود. درخت تبارزایی درختی است که نشاندهندهٔ روابط تکاملی در میان انواع مختلف گونههای زیستی، بر اساس شباهت ژنتیکی آنها است. یک دسته از روشهای مورد استفاده برای یافتن گونههایی که از لحاط ژنتیکی به یکدیگر نزدیک هستند، روشهای خوشهبندی از جمله k-medoids ،k-means و DBSCAN هستند. در بین این روشها، DBSCAN دقت و سرعت بیشتری در خوشهبندی دادههای ژنتیکی دارد.[4] علاوه بر این روشها، از روشهای یادگیری عمیق مانند شبکههای عصبی پیچشی نیز استفاده میشود.[5]
متنکاوی
با افزایش تعداد نشریات زیستشناسی جستجو و جمعآوری اطلاعات در مورد موضوعی خاص به امری دشوار تبدیل شد. به این امر استخراج دانش گفته میشود. جمعآوری اطلاعات از تمامی منابع موجود در مورد دادههای زیستی بسیار مهم است چرا که این اطلاعات در ادامه میتوانند به تولید دانش زیستی جدید با استفاده الگوریتمهای یادگیری ماشین کمک کنند. برای استخراج دانش از گزارشهایی که به دست انسان تولید شدهاند، میتوان از روشهای پردازش زبان طبیعی استفاده کرد.[2]
سایر کاربردها
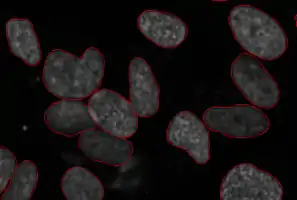
یکی از زیرشاخههایی که روشهای یادگیری ماشین در آن نقش مهمی دارند آنالیز تصاویر زیستی است. در این زیرشاخه به طراحی روشهایی برای آنالیز محاسباتی تصاویر زیستی پرداخته میشود. به صورت مرسوم، آنالیز تصاویر زیستی به دست خود انسان انجام میشود. این روش کند و پرهزینه است و نتیجهٔ آن وابسته به شخصی است که تصاویر را آنالیز میکند. علاوه بر این، میکروسکوپهای خودکار مدرن قادر به تولید صدها تا هزاران تصویر در هر ساعت هستند، که آنالیز دستی این تصاویر را غیرممکن میکند. به همین دلیل از روشهای بینایی ماشین و تشخیص الگو در آنالیز تصاویر زیستی کمک گرفته میشود. یکی از بزرگترین زیرمجموعهها از تصاویر زیستی که به آنالیز خودکار نیاز دارد، تصاویر میکروسکوپهای فلئورسانس است. برای این گروه از تصاویر، مسئلههای بخشبندی سلولها، دستهبندی واکنشهای فنوتیپی و تصمیمات مربوط به واکنشهای مشتق از آن، بهطور معمول مطرح میشوند.[7]
از زیرشاخههای دیگری که در آنها از روشهای یادگیری ماشین استفاده میشود میتوان به طراحی پرایمر، آنالیز دادههای طیفسنج جرمی و ترجمهٔ معکوس پروتئینها اشاره کرد.
موضوعات مرتبط
- ژنومیک
- پروتئومیک
- آنالیز تصاویر زیستی
- ریزآرایه
- زیستشناسی دستگاهها
- تکامل
- بیوانفورماتیک
- یادگیری ماشین
- زیستشناسی محاسباتی
- انفورماتیک پزشکی
منابع
- Larrañaga, Pedro; Calvo, Borja; Santana, Roberto; Bielza, Concha; Galdiano, Josu; Inza, Iñaki; Lozano, José A.; Armañanzas, Rubén; Santafé, Guzmán (2006-03-01). "Machine learning in bioinformatics". Briefings in Bioinformatics. 7 (1): 86–112. doi:10.1093/bib/bbk007. ISSN 1467-5463.
- "Machine learning in bioinformatics". Wikipedia. 2019-07-10.
- «GenBank and WGS Statistics». www.ncbi.nlm.nih.gov. دریافتشده در ۲۰۱۹-۰۷-۲۳.
- Mahapatro, Gayatri; Mishra, Debahuti; Shaw, Kailash; Mishra, Sashikala; Jena, Tanushree (2012). "Phylogenetic Tree Construction for DNA Sequences using Clustering Methods". Procedia Engineering. 38: 1362–1366. doi:10.1016/j.proeng.2012.06.169.
- Schrider, Daniel R.; Hochuli, Joshua; Suvorov, Anton (2019-06-18). "Accurate inference of tree topologies from multiple sequence alignments using deep learning". bioRxiv: 559054. doi:10.1101/559054.
- Coelho, Luis Pedro; Shariff, Aabid; Murphy, Robert F. (2009-6). "Nuclear segmentation in microscope cell images: A hand-segmented dataset and comparison of algorithms". (:unav). doi:10.1109/isbi.2009.5193098. PMC 2901896. PMID 20628545. Check date values in:
|date=(help) - Coelho, Luis Pedro; Glory-Afshar, Estelle; Kangas, Joshua; Quinn, Shannon; Shariff, Aabid; Murphy, Robert F. (2010). Blaschke, Christian; Shatkay, Hagit, eds. "Principles of Bioimage Informatics: Focus on Machine Learning of Cell Patterns". Linking Literature, Information, and Knowledge for Biology. Lecture Notes in Computer Science. Springer Berlin Heidelberg: 8–18. doi:10.1007/978-3-642-13131-8_2. ISBN 978-3-642-13131-8.