دیانای ژیراز
دیانای ژیراز یا به عبارت سادهتر ژیراز، آنزیمی است از دستهٔ توپوایزومرازها که مربوط به زیردستهٔ دوم آنها است که در فرایندهای داخل سلولی مانند همانندسازی رشته دیانای یا رونویسی از دیانای نقش دارد.[1] این آنزیم در یک فرایند وابسته به انرژی آدنوزین تریفسفات(ATP) هنگامی که آرانای پلیمراز یا دیانای هلیکاز در حال بازکردن دیانای دو رشتهای در ناحیههای مخصوص برای شروع همانندسازی به نام مکان آغازش همتاسازی هستند، در پشت آنها روی دیانای قرار میگیرد و باعث کاهش پیچ و تاب خوردگیهای قسمتهایی از رشته دیانای که همانندسازی از روی آنها به اتمام رسیدهاست.[2][3] این آنزیم در واقع وظیفه دارد تا با فشردهسازی و پیچوتاب دادن به رشتهٔ بلند دیانای، آن را در فضای کوچک هسته جا کند که این کار را با ایجاد کردن فشردهسازیهای منفی یا به عبارت دیگر با آزادسازی فشردهشدگیهای مثبت انجام میدهد. این فرایند فشردهسازی در سلولهای پروکاریوتها به خصوص باکتریها اتفاق میافتد زیرا که رشتهٔ دیانای این موجودات به صورت یک حلقه دایرهای است و دیانای ژیراز با قطع کردن این رشته دایرهای، فشردهسازی که باعث تغییر در تعداد پیچوتابهای رشته دیانای میشود را انجام میدهد و در انتهای کار دو سر جدا از هم دیانای را به هم میپیچاند. این خاصیت آنزیم دیانای ژیراز که باعث به وجود آمدن فشردگیهای منفی یا تبدیل کردن فشردگیهای مثبت به منفی است، این اجازه را به دیانایی باکتری میدهد که به سادگی از روی آن همانندسازی و رونویسی انجام شود زیرا که به صورت طبیعی رشته دیانای پیچشهای مثبت پیدا میکند، حال این آنزیم جلوی این امر را میگیرد و امکان جلو رفتن دیانای هلیکاز بر روی رشته دیانای فراهم میسازد تا همانندسازی یال رونویسی به صورت کامل تکمیل شود. آنزیم دیانای ژیراز غالباً در کلروپلاست گیاهان مختلف یافت میشود.[1][4]
نمونهٔ باکتریایی دیانای ژیراز هدف نابودسازی بسیاری از آنتیبیوتیکها از جمله اسید نالیدیکسیک، نووبیوسین و سیپروفلوکساسین است.
supercoils2.png.webp)
ساختار دیانای ژیراز
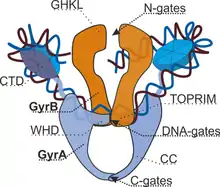
آنزیم دیانای ژیراز از دسته آنزیمهای تترامتریک است؛ بدین معنا که ساختار آن از ۴ بخش گوناگون تشکیل شدهاست. این ساختار شامل دو بخش GyrA (یا زیربخشهای A) و دو بخش GryB (یا زیربخشهای B) است.
ساختار پیچیدهٔ آنزیم دیانای ژیراز مخلوطی از سه جفت دریچه(gate) به نامها N-gates, C-gates و DNA-gates، نواحی ورودی و خروجی که رشته دیانای در فاصلهٔ بین آنها منتقل میشود و ناحیهای که فشردگیهای منفی در آن به وجود میآیند. دریچههای N-gates توسط بخش GyrB ساخته شدهاند و با متصل شدن دو مولکول ATP به آنها و در نتیجه آزاد کردن انرژی، باعث شروع شدن فرایندهای بین آنزیم و رشتهٔ دیانای که به آن متصل شده میشوند. فرایند هیدرولیز در جهت عکس باعث بسته شدن دریچهها میشود. شکافته شدن و بسته شدن رشته دیانای که حاصل یک سری فرایندهای کاتالیستی است که در DNA-gates اتفاق میافتد، این دریچهها از همهٔ بخشهای کل آنزیم تشکیل شدهاند یعنی GyrA و GyrB. دریچههای C-gates از قسمتهای GyrA آنزیم ساخته شدهاند.[5]
نحوهٔ عملکردن آنزیم ژیراز
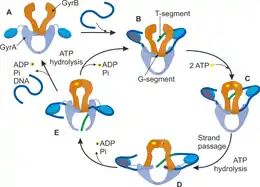
در هنگامی که دیانای ژیراز به رشتهٔ دیانای متصل میشود، همانطور که در قسمت قبل به آن اشاره کردیم دو مولکول ATP که شامل بستههای انرژی هستند به ساختار آنزیم میچسبند و باعث تغییر شکل در قسمت GyrB شده و در نتیجه قسمت جابهجا شوندهٔ رشته DNA در بین زیربخشهای قسمت GyrB آنزیم قرار میگیرد. در مرحله بعد قسمت gate-segment دیانای (در شکل نشان داده شدهاست) در DNA-gates آنزیم قرار میگیرد و که باعث پارهشدن هر دو رشتهٔ دیانای میشود. سپس با هیدرولیزه شدن یکی از مولکولهای ATP متصل شده، قسمت جابهجا شونده دیانای (در شکل با عنوان T-segment نمایش داده شدهاست) از بین T-segment که گسسته شدهاست، عبور میکند. در انتها آنزیم ناحیهٔ پاره شدهٔ رشته دیانای یعنی G-segment را پیوند میزند و در انتها T-segment از ساختمان آنزیم دیانای ژیراز خارج میشود. با هیدرولیزه شدن مولکول ATP دوم، ساختار آنزیم به حالت اولیهٔ خود بازمیگردد.[6]
در نتیجه در هر چرخه کاتالیستی که آنزیم ژیراز طی میکند، دو مولکول ATP هیدرولیز میشوند و دو فشردگی منفی به رشتهٔ دیانای که فرایند بر روی آن انجام شدهاست، اضافه میشود. بر اساس مشاهدات میتوان به این نتیجه دست یافت که به ازای هر دو مولکول ATPای که در هر چرخه از فعالیت آنزیم هیدرولیز میشوند، تعداد پیچخوردگی مثبت در طول رشتهٔ دیانای دو واحد کاهش پیدا میکند.[7]
اثر آنتیبیوتیکها بر ژیراز
آنزیم ژیراز در سلولهای پروکاریوتها و برخی از یوکاریوتها وجود دارد، اما بدین شکل نیست که آنزیم ژیراز در تمام سلولهای این موجودات کاملاً یکسان باشند. این آنزیم در سلولهای مختلف دارای ساختار و توالی پروتئینی متفاوتی است که این خاصیت سبب میشود تا هدف مناسبی برای آنتیبیوتیکها باشد. برخی از آنتیبیوتیکها میتوانند به صورت خاص بخشهای خاصی از ساختمان آنزیم ژیراز یعنی قسمتهای gyrA و gyrB را مختل کنند مانند اسید نالیدیکسیک.[8] اما دو دسته از مهمترین آنتیبیوتیکهایی که اثر بازدارندگی بر روی آنزیم ژیراز دارند به شرح زیر است:
- آمینوکرامینها. این آنتیبیوتیکها با متصل شدن به ناحیهای از آنزیم که ATPها بر آن قرار میگرفتند (دو زیرقسمت GryB)، چرخهای که آنزیم بر اساس آن کار میکرد را مختل میکنند و در نتیجه آنزیم دیگر قادر به فعالیت نخواهد بود.
- فلوروکینولونها. از این آنتیبیوتیکها به عنوان قاتل توپوایزومرازها یاد میشود. این دسته از آنتیبیوتیکها با متصل شدن به ساختمان آنزیم، فضای بین DNA-gates را اشغال میکنند و از پیوند خوردن دو رشته دیانای که از همدیگر گسسته شده بودند، جلوگیری میکند و در نتیجه در چرخهٔ فعالیت آنزیم اختلال ایجاد میکند. این فرایند باعث میشود که رشته دیانایهای پاره شده در درون سلول انباشته شوند و همانندسازی دیانای به درستی شکل نگیرد و به مرگ سلول منجر میشود.
جستارهای وابسته
- توپوایزومرازها
- آنزیم
- آمینواسیدها
- توالی پروتئین
- دیانای هلیکاز
- فشردهسازیهای منفی
- فشردهشدگیهای مثبت
پانویس
- Garrett RH, Grisham CM (2013). Biochemistry (5th, International ed.). United States: Mary Finch. p. 949.
- Wigley, D. B.; Davies, G. J.; Dodson, E. J.; Maxwell, A.; Dodson, G. (1991-06-20). "Crystal structure of an N-terminal fragment of the DNA gyrase B protein". Nature. 351 (6328): 624–629. doi:10.1038/351624a0. ISSN 0028-0836. PMID 1646964.
- Morais Cabral, J. H.; Jackson, A. P.; Smith, C. V.; Shikotra, N.; Maxwell, A.; Liddington, R. C. (1997-08-28). "Crystal structure of the breakage-reunion domain of DNA gyrase". Nature. 388 (6645): 903–906. doi:10.1038/42294. ISSN 0028-0836. PMID 9278055.
- Evans-Roberts, Katherine M.; Mitchenall, Lesley A.; Wall, Melisa K.; Leroux, Julie; Mylne, Joshua S.; Maxwell, Anthony (2016-02-12). "DNA Gyrase Is the Target for the Quinolone Drug Ciprofloxacin in Arabidopsis thaliana". The Journal of Biological Chemistry. 291 (7): 3136–3144. doi:10.1074/jbc.M115.689554. ISSN 1083-351X. PMC 4751362. PMID 26663076.
- Bush, Natassja G.; Evans-Roberts, Katherine; Maxwell, Anthony (2015). "DNA Topoisomerases". EcoSal Plus. 6 (2). doi:10.1128/ecosalplus.ESP-0010-2014. ISSN 2324-6200. PMID 26435256.
- Basu, Aakash; Parente, Angelica C.; Bryant, Zev (05 08, 2016). "Structural Dynamics and Mechanochemical Coupling in DNA Gyrase". Journal of Molecular Biology. 428 (9 Pt B): 1833–1845. doi:10.1016/j.jmb.2016.03.016. ISSN 1089-8638. PMC 5083069. PMID 27016205. Check date values in:
|date=(help) - Reece, R. J.; Maxwell, A. (1991). "DNA gyrase: structure and function". Critical Reviews in Biochemistry and Molecular Biology. 26 (3–4): 335–375. doi:10.3109/10409239109114072. ISSN 1040-9238. PMID 1657531.
- Engle, E. C.; Manes, S. H.; Drlica, K. (1982-1). "Differential effects of antibiotics inhibiting gyrase". Journal of Bacteriology. 149 (1): 92–98. ISSN 0021-9193. PMC 216595. PMID 6274849. Check date values in:
|date=(help)